Nodファクター受容体であるNFR1は、キチンを受容して防御応答を発動させるCERK1と非常によく似ています。その上で私の研究により、NFR1には防御応答を起動する能力があることが判明しました。

NFR1もCERK1も、細胞外でそれぞれNodファクターやキチンを受容した後で、細胞内のキナーゼドメインという他のタンパク質をリン酸化する部分が働くことで、共生応答や防御応答が起動すると考えられています。

NFR1がペアを組むNFR5もLysM型受容体キナーゼですが、構造的にも実験的にもキナーゼ活性が無いことが証明されていますし、ミヤコグサのNFR1およびシロイヌナズナのAtCERK1では、それぞれの細胞内キナーゼドメインの活性が、下流の応答の起動に必須であることが証明されています。キナーゼ部分(青字)のアミノ酸配列を比較したところ、下のように非常に類似していることが判りました(パソコン推奨です。すいません)。キナーゼには多くの種類があって多少は似ているのですが、ここまで相同性が高いのは異様です。

もしかするとAtCERK1のキナーゼドメインは、実質的にNFR1と同じなのでしょうか??
ミヤコグサのnfr1変異体は、全く根粒菌が感染できません。ここに正常なNFR1を導入する「相補実験」を行うと、当然ながら機能が回復して共生できるようになります。それではNFR1の代わりにAtCERK1を入れるとどうなるでしょうか? さすがに細胞外の部分がキチンに対応しているのでダメだと思うのですが、細胞外がNFR1で細胞内がAtCERK1では・・・?
そこで実際に試してみることにしました。
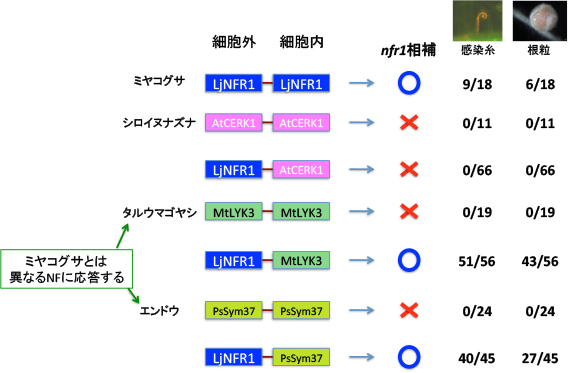
AtCERK1をそのままミヤコグサnfr1変異体に導入しても共生機能は回復しませんが、細胞外をNFR1に代えてNodファクターに応答できるようにしてもダメでした。したがってアミノ酸配列が類似していても、やっぱりAtCERK1とNFR1は違うようです。ついでに異なるNodファクターに応答するタルウマゴヤシやエンドウのNFR1を使って、同様の実験をしてみました。この場合、細胞外ドメインをミヤコグサのLjNFR1にしてやると、見事に相補します(LjNFR1-MtLYK3, LjNFR1-PsSym37)。この結果から、
- 異なる植物種の遺伝子の細胞外ドメインと細胞内ドメインをキメラにすることは問題では無い。
- 異なる植物のNFR1細胞内ドメインもミヤコグサで機能する
ということが確認できました。つまりこの実験自体には問題が無く、AtCERK1の細胞内ドメインのアミノ酸配列に共生応答を誘導する機能が無いということです。

コメント