NFR1とAtCERK1のキナーゼは、アミノ酸配列が類似していますが、NFR1だけに共生応答を起動する能力がありました。
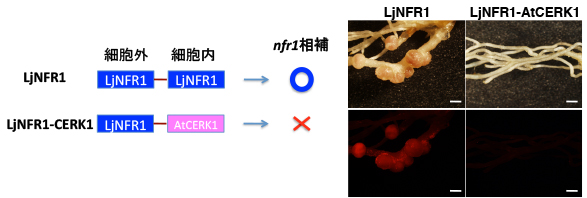
ということは、NFR1の共生能力はAtCERK1とのわずかな違いで決まっているはずです。そこでNFR1の配列を部分的にAtCERK1に置き換えて、どこを置換した場合に共生能力が失われるかを調べました。
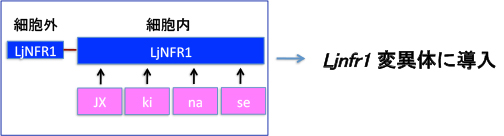
共生に必須の領域を絞り込んで行きます・・・・
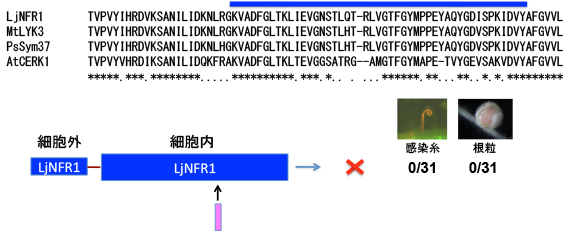
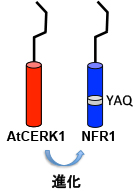
そして最終的には、3アミノ酸のYAQという配列が決定的な役割を果たしていることが判りました。この部分はAtCERK1ではTVというアミノ酸配列になっています。
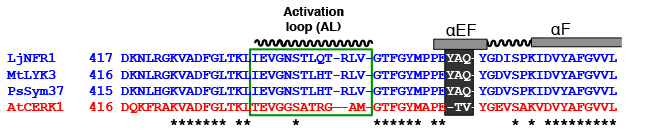
このTV配列を下記のようにYAQに換えると、見事に全く共生機能が無かったNFR1-AtCERK1が共生応答を起動できるようになりました!!
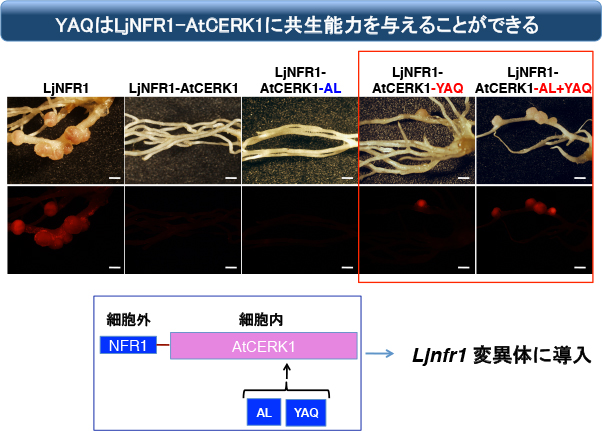
ちなみにYAQ単独だと共生機能は非常に弱いのですが、Activation Loop(AL)の配列もNFR1型にすると、かなり多くの根粒が形成されます。しかしALだけを与えても共生機能は無いので、YAQが根本的に重要であることが判ります。
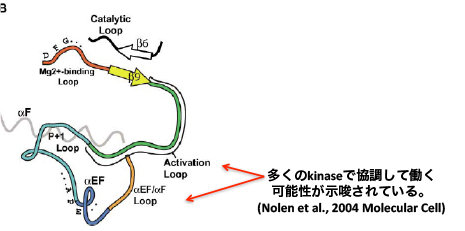
ALはもともとキナーゼの活性に重要であることが多いのですが、YAQはALに影響する可能性が指摘されているαEFヘリックスという構造を作ります。YAQはALの構造に干渉することで、リン酸化する強さや標的の種類などに影響を与えるのでしょう。
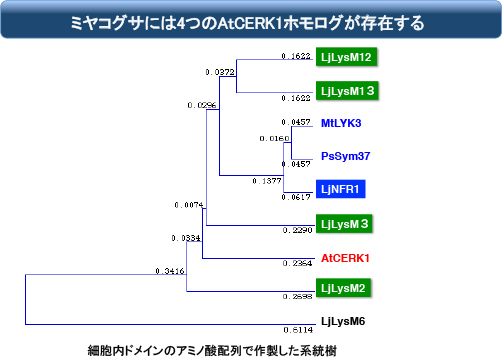
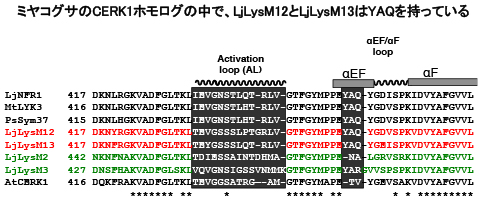
ちなみにミヤコグサには、NFR1/CERK1に似た遺伝子が他に4つあります。その中の2つはYAQ配列を持っています。
NFR1が壊れた時点で根粒菌共生が完全に破綻するので、他の4つは少なくともそのままでは機能しないですが、それは細胞外ドメインがミヤコグサ根粒菌に適合していないだけかもしれません。そこで細胞外をNFR1に換えて同様の実験を行うと・・・
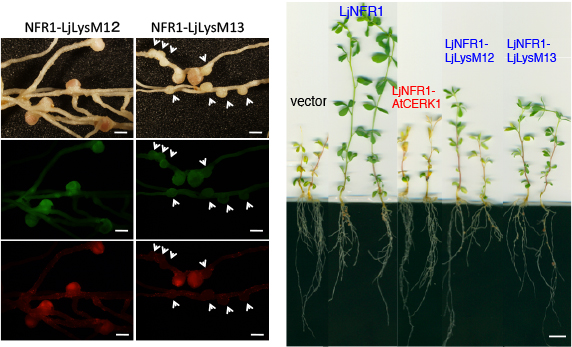
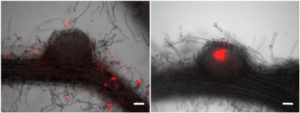
面白いことに根粒はできるのですが、感染が途中で止まってしまうものが多く完全には相補できないことが判りました。どうやら「完全な根粒菌共生を誘導する能力は無いが・・・」という中途半端な感じです。
以上の結果から、おそらくはマメ科の祖先植物で防御応答に機能していたCERK1が、YAQ配列を獲得して根粒共生が可能になった。その後にALをさらに改変して効率を高めてたのだろう・・・と、この時は考えていました。マメ科だけにNFR1/CERK1型のLysM型受容体キナーゼが多いのは、NFR1を産み出すための試行錯誤の痕跡かもしれません(LysM12, 13はその苦労の跡?)。
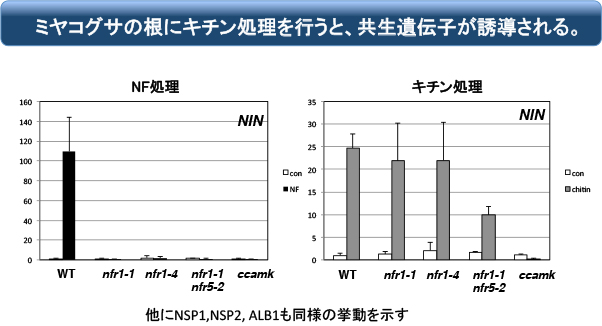
面白いことにミヤコグサにキチンを処理して応答する遺伝子の中には、共生遺伝子が含まれます。これはnfr1変異体でも観察されるので、マメ科の祖先が持っていたCERK1には、共生応答を誘導する素養があったのでしょう(と、考えていました)。
ここまでの成果をまとめて、責任著者として2011年に論文発表したのですが、これまでに100回以上引用されており、今年も既に14報の論文で言及されています。新規遺伝子の報告ではない論文で、この引用数は悪くない数字だと考えています。


コメント