根粒で高発現するGmPEPC7と全ての組織で弱く発現するGmPEPC15は、そのプロモーター領域に差があると推測されます。そこでGmPEPC7のプロモーターを少しずつ改変して、それをダイズに導入して根粒での発現を調べる戦略を私達は採用しました。

上の図はmRNAが転写される最初の位置を+1番目のDNAとした時のGmPEPC7とGmPEPC15の配列を比較したものです(パソコン推奨です。すいません・・・)。-600bpまで非常に高い保存性を示しています。この部分に「根粒でRNAを大量に作製する」という命令が書かれているかもしれません。そこで、この領域のDNA断片の下流にGUSという遺伝子を接続したものをダイズに形質転換しました。
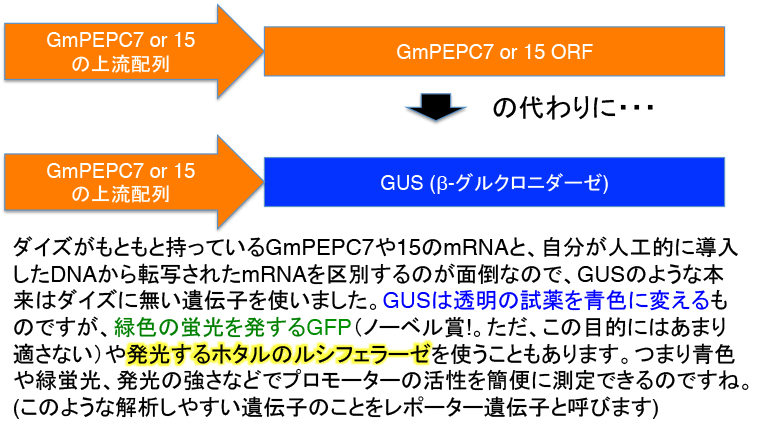
GUSという遺伝子は大腸菌の遺伝子で、この酵素活性は植物にはほとんどありません。ある試薬を与えて反応させると、GUSが発現している部分が青く染まるので解析しやすいのです。下図のA-CはGmPEPC7の-1352bpまでのDNA断片を接続したものですが、根粒が青く染まっています。一方でGmPEPC15を接続したものは活性がほとんどありません(D)。
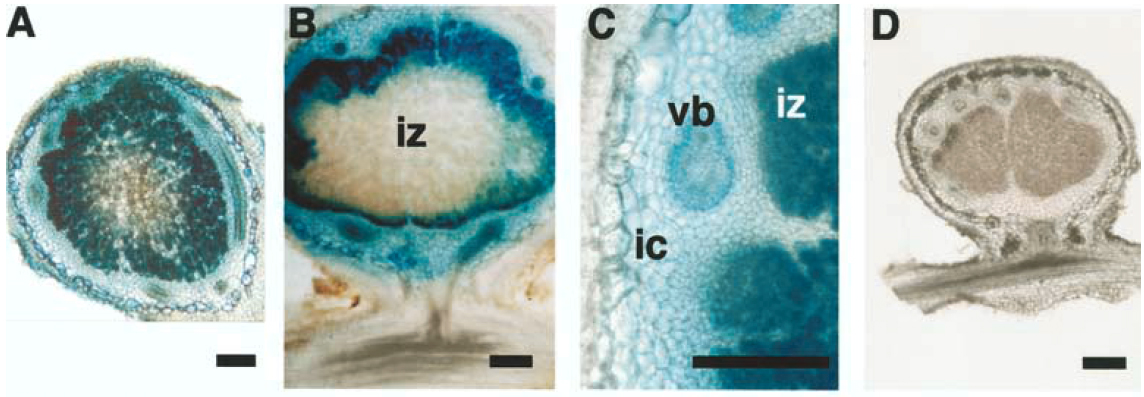
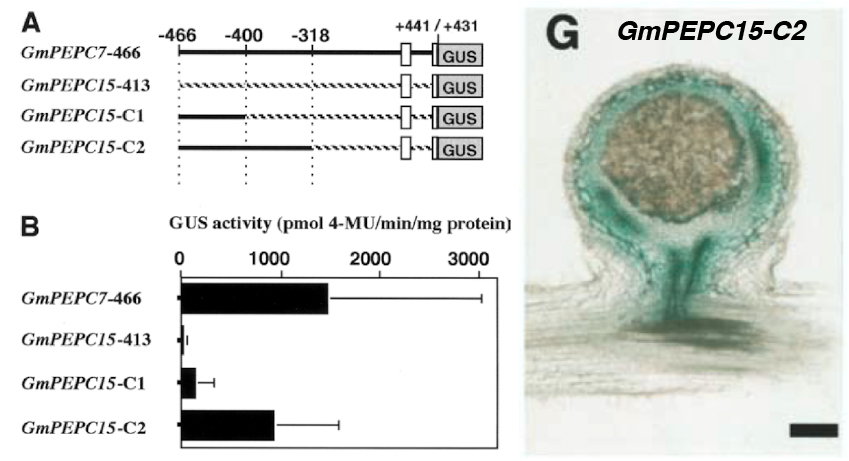
次にGmPEPC15と保存性の高い領域(-466まで)のみを使って解析したところ、下記のようにGUSの強い活性が検出されましたが、-400までにすると活性が消えてしまいました。-466から-400の領域をGmPEPC15の対応する領域と入れ替えたGmPEPC7-C1も活性がありません。したがってGmPEPC7の-466から-400の領域に、根粒で高く発現するために重要な配列があると推測されます。
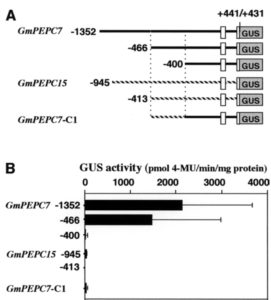
核でRNAが転写される時には、プロモーター領域の特定の配列に転写因子と呼ばれるタンパク質が結合して転写を活性化(または阻害)します。そこでGmPEPC7の-466から-400のDNA領域(SR領域と後で名付けました)に結合するタンパク質を検出することを試みました。ダイズ根粒の核に含まれるタンパク質を抽出して、それにDNA断片を混ぜて電気泳動します。仮にDNA断片にタンパク質が結合すれば、本来の移動度よりも大幅に遅れて電気泳動されるでしょう(このような実験をゲルシフトアッセイと言います)。
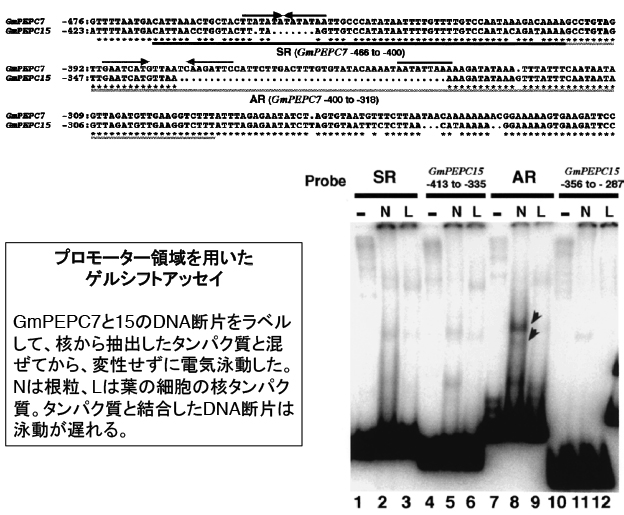
しかし、SR領域に結合するタンパク質はほとんど検出されませんでした。一方でSR領域のすぐ下流(AR領域と後で名付けます)は、GmPEPC7だけに結合する根粒の核タンパク質が検出されました。AR領域はGmPEPC15で大きく欠損しています。したがってAR領域も何か役割があるかもしれません。
そこでGmPEPC15のプロモーター領域にSRとARをそれぞれ移植したところ、SRだけでも弱い活性を示しましたが、SRとARを両方とも移植すると強烈な活性を示すことが判りました。ちなみに上の方の図で、ARしか持たないGmPEPC7-C1に活性が無いことが判明していますので、SRは根粒での発現に必須で、ARがそれを増幅するような役割を果たしていると推測されます。
以上の結果からGmPEPC7は、祖先の根粒共生とは関係の無かった遺伝子がSRとARを獲得したことで根粒特異的な高発現をするようになって進化したと考えられます。おそらく最初にSRを獲得したことで根粒である程度機能するようになり、後でARを獲得してさらなる大量発現を実現したのでしょう。
ただ・・・GmPEPC15に関してはGmPEPC7の祖先遺伝子であるというシナリオ以外に、GmPEPC7が先に誕生してから派生した遺伝子である可能性(つまり後から機能を失った)を否定できません。どちらにせよ、発現制御機構が変化することで、根粒共生に必要な遺伝子が獲得されうることを示すことができたと考えています。(Mol. Gen. Genomics 269: 163-172, 2003)


コメント